0-SAM2-UNet: Segment Anything 2 Makes Strong Encoder for Natural and Medical Image Segmentation
SAM2 - UNet:利用 Segment Anything 2 为自然和医学图像分割打造强大编码器总结

arXiv:2408.08870v1 [cs.CV] 16 Aug 2024
背景
图像分割是计算机视觉重要任务,多种下游任务依赖于它,但设计统一架构处理不同分割任务仍是挑战。视觉基础模型(VFMs)如 SAM2 在图像分割有潜力,但存在生成与任务无关的分割结果等问题,需探索提升其适应性和性能的策略。已有研究尝试将 SAM 适配到下游任务,如使用适配器、集成额外输入或改变架构,但受限于 SAM1 中 ViT 编码器的结构,SAM2 的分层骨干为设计更有效的 U 形网络提供了新机会。
所以作者提出 SAM2 - UNet,证明 Segment Anything Model 2(SAM2)可作为 U 形分割模型的强大编码器,通过简单有效的框架实现通用图像分割。
实验方法
模型架构
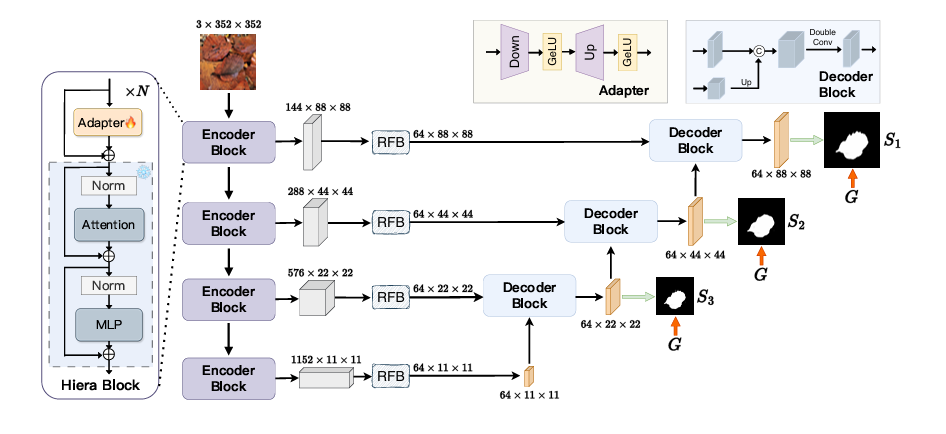
SAM2-UNet 概述。Hiera 块有一些变体,为了便于理解,只演示了一个简化的结构。
编码器
采用 SAM2 预训练的 Hiera 骨干网络,其分层结构适合 U 形网络,能多尺度捕捉特征,输出不同层次特征。
感受野模块(RFBs)
用于减少编码器特征的通道数至 64 并增强特征。
适配器
因 Hiera 参数量大,冻结其参数并在每个多尺度块前插入适配器,实现参数高效微调,适配器由线性层、GeLU 激活函数等组成。
解码器
采用经典 U - Net 设计,包含三个解码器块,每个块有两个 “Conv - BN - ReLU” 组合,输出特征经 1×1 Conv 分割头生成分割结果并上采样,与真实掩码监督训练,使用加权 IoU 损失和二元交叉熵(BCE)损失作为训练目标并应用深度监督。
实验结果
在五个不同基准上的十八个数据集进行实验,包括伪装对象检测、显著对象检测、海洋动物分割、镜子检测和息肉分割任务,各任务采用相应数据集和评估指标。
基于 PyTorch 在单张 NVIDIA RTX 4090 GPU 上实现,使用 AdamW 优化器,采用随机垂直和水平翻转的数据增强策略,输入图像调整为 352×352,不同任务设置不同训练轮数,息肉分割任务采用多尺度训练策略。
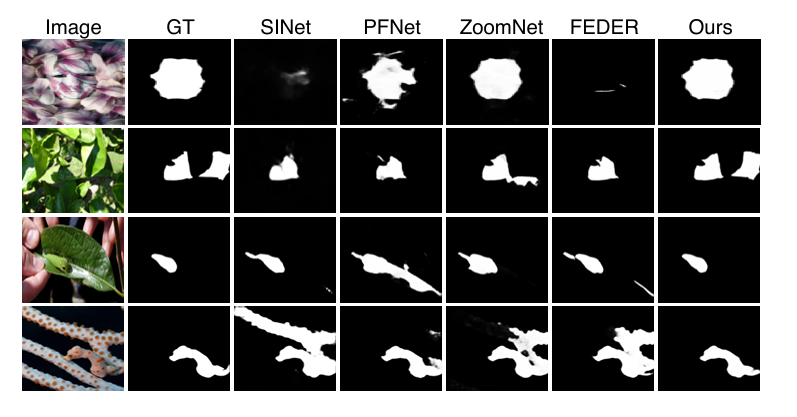
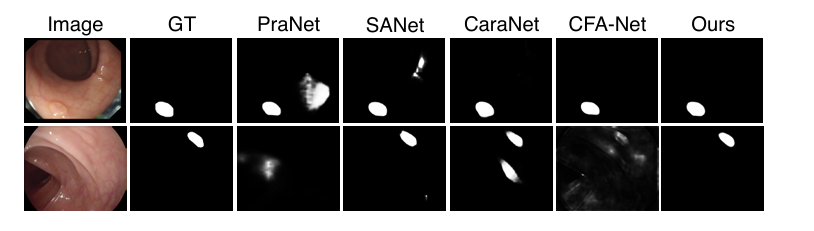
在伪装对象检测、显著对象检测、海洋动物分割、镜子检测和息肉分割任务中,SAM2 - UNet 在多个数据集上的各项评估指标上表现优异,超过其他对比方法,部分任务实现了最先进性能,可视化结果也显示其在不同场景下的准确性优势。
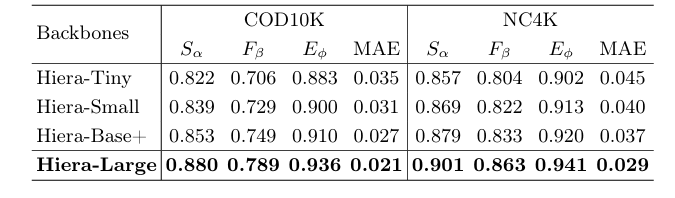
消融实验
对 Hiera 骨干网络大小进行消融实验,结果表明较大骨干通常性能更好,但较小骨干的 SAM2 - UNet 也能取得不错结果,证明 SAM2 预训练的 Hiera 骨干网络提供的高质量表示有效。
总结
提出的 SAM2 - UNet 框架简单有效,适用于自然和医学图像分割任务,其采用的 SAM2 预训练 Hiera 编码器与经典 U - Net 解码器结合的方式被证明有效,可作为未来 SAM2 变体开发的新基线。
代码分析
使用了SAM2.0基础模型的编码器与解码器,去除了其提示模块,并构建了无零样本分割能力的网络。
1 | |
通过设置不同的上、下采样数值,在前项传播中进行多尺度的融合计算。通过插值法bilinear计算3个不同的输出。
1 | |
感受野模块用来确保尺度对齐。